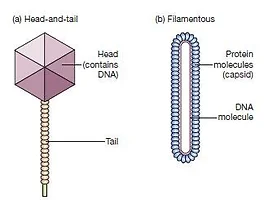
모든 내용은 Gene Cloning and DNA Analysis, 6th edition, T.A.Brown, Wiley-Blackwell을 참고하여 작성하였습니다.
본 글의 제목은 언급된 도서의 각 챕터의 절 이름과 동일합니다.
대부분의 그림 역시 본 책에서 왔으며, 그 이외에는 따로 표시합니다.
도움이 되었다는 댓글 하나가 큰 힘이 됩니다!
Chapter 3
Purification of DNA from Living Cells
생명공학에서 얻어내고자 하는 주요 DNA 종류
① Total cell DNA: genomic DNA (chromosome) + any additional DNA (plasmid)
② Plasmid DNA
③ Phage DNA
3.1 Preparation of total cell DNA
Chromosome DNA나 plasmid DNA 등을 구분하지 않고 한꺼번에 얻어냄
Total DNA preparation을 위한 4단계
① A culture of bacteria is grown and then harvested.
② The cells are broken open to release their contents.
③ This cell extract is treated to remove all components except the DNA.
④ The resulting DNA solution is concentrated.
① Growing and harvesting a bacterial culture
대부분의 세균들은 액체 배지(liquid medium, broth)에서 잘 배양됨(broth culture)
Defined medium: 모든 성분과 그 양이 밝혀진 배지. 정밀하게 조절된 환경이 필요할 때 씀
Undefined medium: 성분 조성과 그 양이 대략적으로만 알려진 배지. 일반적인 배양에 사용
LB(Luria-Bertani) medium의 경우 yeast extract를 tryptone으로 처리하고 NaCl을 첨가한 것이 성분의 전부
E. coli를 LB에서 배양 시, 37℃에서 150-250rpm으로 회전시켜 공기를 공급해주면서 배양함
세균이 많이 자랄수록 배지는 뿌옇게 흐려지며 이에 따라 배지의 흡광도(optical density, OD)도 달라지므로 OD 측정을 통해 배지 내의 세균 밀도를 알 수 있음
OD = log10(통과한 빛의 세기 / 조사한 빛의 세기)
600nm 가시광선 기준으로 OD=1일 때 E. coli 농도는 대략 0.8×109 cells/ml
전체 배지 내에 매우 적은 양을 차지하는 DNA를 얻어내기 위해서는 cell을 농축시키는 과정이 필요
Centrifugation 하여 배지 내의 cell들을 모두 바닥의 pellet 형태로 가라앉힘
대량으로 세포를 배양했을 경우 8000rpm 정도, 소량일 경우 13200rpm 정도의 빠르기로 centrifugation
② Preparation of a cell extract
Bacterial cell은 cell wall과 cell membrane을 가지므로 이것을 먼저 제거해야 내부의 DNA를 꺼낼 수 있음
Physical method: Sonication. DNA는 손상되지 않는 영역의 음파를 이용해 세포벽과 세포막을 부숨.
많은 열이 발생하므로 항상 cell이 담긴 용기를 얼음 속에 넣은 채로 실행해야 함
Chemical method: 화학 물질을 사용해 세포벽과 세포막을 불안정하게 만들고 구조를 파괴
Lysozyme은 peptidoglycan cell wall의 1,4-beta linkage를 파괴
EDTA는 세포벽과 DNA에 결합한 Mg2+를 chelating하여 세포벽을 불안정화시키고 DNA를 좀 더 순수한 형태로 얻을 수 있게 해 줌
SDS(sodium dodecyl sulfate)는 계면활성제로 세포막을 파괴하고 단백질을 변성시킴
이런 방법들을 사용한 이후 배지를 다시 원심 분리하여 세포 잔해들을 가라앉히고 DNA가 든 상층액만을 분리함
Lysozyme, EDTA → cell wall
SDS → protein,cell membrane
③ Purification of DNA from a cell extract
분리된 상층액 속에는 DNA뿐만 아니라 RNA와 protein도 일부 포함됨
이것들을 제거하기 위해 DNA purification을 시행
Phenol extraction: Protease로 protein을 작게 자른 후 phenol과 섞어 protein을 엉겨붙게 하여 DNA와 RNA를 순수하게 분리. RNA를 제거하는 작업이 추가로 필요(ribonuclease 이용)하고 phenol이 독성 물질이기 때문에 잘 쓰지 않음
Ion-exchange chromatography: (+) resin(or bead) column에 DNA, RNA, protein이 혼합된 상층액을 흘려 넣으면 이 중 음전하를 가장 많이 띠는 DNA가 resin에 가장 잘 붙는 점을 이용. 농도가 다른 NaCl solution을 순차적으로 넣어 charge 정도에 따라 column에서 분리할 수 있음(fraction collector). 굉장히 비싼 편.
두 방법 모두 몸에 좋지 않거나 비싸기 때문에 최근에는 간편하고 저렴한 silica gel이 널리 이용됨
④ Concentration of DNA samples
얻어낸 DNA의 농도가 낮을 경우 농축을 통해 농도를 높일 수 있음
Ethanol precipitation: Ethanol이 물 분자와 수소결합을 이루면 상대적으로 DNA가 물과 분리되는 것을 이용. Na+와 같은 1가 양이온의 존재 하에 100% ethanol을 –20℃ 이하의 온도에서 DNA의 2.5배 정도로 넣으면 DNA가 빠르게 흰 실처럼 엉키는데, 이 때 유리막대를 넣어 돌리면 막대에 DNA가 붙어 나옴
이 방법은 보통 긴 chromosomal DNA에 사용하며, plasmid DNA는 양이 적고 잘 엉겨 붙지 않으므로 ethanol을 넣고 centrifugation을 함
Phenol extration을 하여 RNase 처리를 했을 경우, RNase에 의해 잘려 나온 ribonucleotide들은 이 과정에서 DNA와 분리됨
Measurement of DNA concentration
Gene cloning을 할 때는 vector와 target gene의 개수 비를 맞춰야 하므로 DNA 농도를 아는 것이 중요함
UV absorbance spectrophotometry를 이용해 DNA의 농도를 측정
260nm에서 흡광도 A260이 1일 때 dsDNA가 50ug/ml의 농도로 존재 (ssDNA는 33ug/ml 정도)
280nm에서는 protein, 230nm에서는 RNA, 330nm에서는 불순물의 농도를 잴 수 있음
A260/A280≥1.80, 즉 DNA가 단백질의 1.8배 이상 존재할 때 purification이 잘 되었다고 할 수 있음
Other methods for the preparation of total cell DNA
세포 유형에 따라 DNA preparation 단계가 달라질 수 있음
동물 세포는 세포벽이 없으므로 detergent 처리만 해도 세포를 파괴할 수 있음
식물 세포는 세포벽 조성이 세균과는 완전히 다르므로 얼린 후 mortar와 pestle에 섞어 갈아 세포벽을 부숨
또한 phenol extraction으로는 식물 속의 많은 탄수화물을 제거할 수 없으므로 cetyltrimethylammonium bromide(CTAB)라는 detergent를 사용해서 DNA-CTAB complex를 만들어 다른 물질로부터 DNA를 분리함
Ion-exchange chromatography는 모든 종류의 DNA 정제에 유용
Silica gel을 쓰는 방법 역시 모든 종류의 DNA에 이용할 수 있고 이 때 사용되는 guanidinium thiocyanate가 핵산을 제외한 모든 물질을 분해하기 때문에 굉장히 편리함
GT 존재 하에 DNA가 silica gel에 붙고 (나머지는 column 통과) 물로 씻어내면 DNA가 쉽게 떨어져 나감
'studio > scienza' 카테고리의 다른 글
| [Chapter 3] Preparation of total cell DNA (3) | 2015.04.12 |
|---|---|
| [생명과학/실험] Inhibiting the action of catechol oxidase (0) | 2015.04.04 |
| [Chapter 2] Bacteriophages (10) | 2015.03.16 |
| [Chapter 2] Plasmids (6) | 2015.03.14 |
| [Chapter 1] What is gene cloning and PCR? Why gene cloning and PCR are so important (6) | 2015.03.14 |